效应子预测的原理及过程 | 技术专栏
一直以来,植物病原菌与寄主植物间的互作研究是植物病理学研究的热点,其相互作用机制存在多种假说,包括模式警戒假说1、诱饵假说2等,表明人们对植物病原菌与寄主植物间互作机制认识的不断完善。寄主植物通过进化出识别效应子的免疫受体并产生效应子触发的免疫反应而对效应子具有抗性。病原菌为了提高适合度和传播能力会产生新的效应子促进其侵染植物寄主,从而产生植物与病原菌的竞争循环。因而效应子可以作为植物防治病害的重要靶标。
目前效应子广义上包括蛋白质、糖类、次级代谢产物等可能参与侵染寄主过程的所有分子3,狭义上将效应子定义为一类利于病原物侵染的蛋白质,其研究较多于卵菌、植物病原细菌和真菌中。特别是植物病原真菌分泌效应子后,需要将效应子转运至准确的寄主亚细胞组分才能发挥其功能,一个主要特征是效应子N末端含有约15-30个疏水氨基酸残基组成的信号肽,因此是否具有信号肽可以作为预测效应子的一个指标。
随着效应子研究的增多,研究人员基于已知效应子提炼出效应子的特征4,主要包括:具有信号肽、无跨膜区域、不高于300个氨基酸、富含半胱氨酸、具有已知的效应子基序或细胞核定位信号、具有内部重复序列、与已知蛋白功能无同源性等。另外与吸器等专化的侵染结构中高表达基因也可以作为候选效应子。利用这些特征我们可以在基因组中预测效应子,减少需要进行功能验证的候选基因数目。
效应子是植物病原物和寄主植物互作中的关键物质,研究效应子有助于了解植物抗病分子机制。而基于转录组测序的效应子预测将有助于我们筛选可能的效应子,缩小功能验证范围,从而更好的研究病原物与植物互作机制,为生物防治奠定理论基础。
现在基于效应子特征,如具有信号肽,无跨膜区域等特性我们可以进行狭义效应子(效应蛋白)的预测,有助于筛选和病原菌与寄主互作相关的基因或蛋白。
那么我们如何进行效应子的预测呢?我们可以使用以下四款软件进行效应子的预测5, 6,分别是SignalP(预测信号肽)7、TMHMM(预测跨膜区)8、TargetP(预测真核蛋白亚细胞定位)9和PredGPI(预测GPI锚定蛋白)10。除了PredGPI只有在线版之外,其余三款软件都有本地和在线两种版本,现在以网页版介绍一下使用方法。
SignalP
在线网址:
http://www.cbs.dtu.dk/services/SignalP/
基本步骤:
1.输入或上传fasta格式的蛋白序列;
2.选择物种:真核生物、革兰氏阴性菌、革兰氏阳性菌。革兰氏阳性菌对应于NCBI分类中的放线菌( Actinobacteria)和厚壁菌(Firmicutes),其他细菌为革兰氏阴性菌;
3.输出格式:默认为标准模式,输出结果图表。如果提交大量序列建议选择“Short”模式。
选择完毕后,点击submit,经过一段时间等待之后会输出结果,结果示例及参数解释如下:
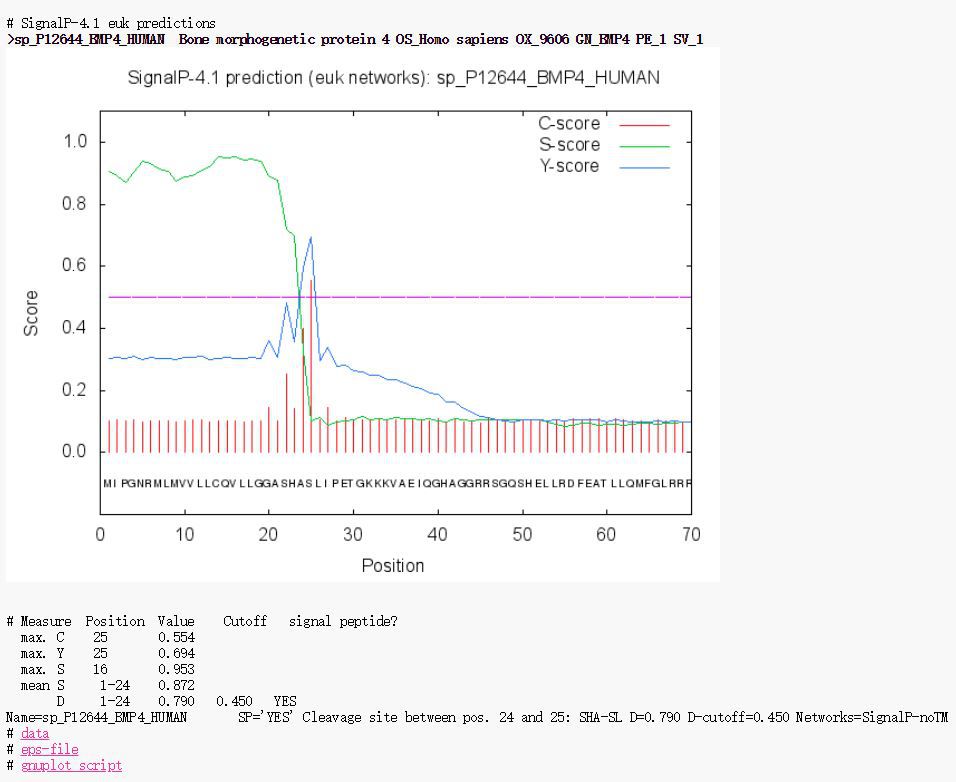
C-score (raw cleavage site score):用来区分是否为剪切位点,最高峰值为剪切位点后的第一个氨基酸(即成熟蛋白的第一个氨基酸残基);
S-score (signal peptide score):用来区分相应位置是否为信号肽区域;
Y-score (combined cleavage site score):C-score和S-score的几何平均数,用于避免多个高分C-score值对结果的影响;
mean S:从氨基酸N端到剪切位点处各氨基酸的S-score平均值;
D-score(discrimination score):mean S和Y-max的加权平均值。
如图在第24-25位点处,C-score和Y-score达到了最高峰,而S-score 发生了陡降,表明在1-24处为预测信号肽区,剪切发生在随后的位点。对应SP=‘YES’表明该蛋白中包含信号肽序列,信号肽剪切位点位于第24、25残基处。
TMHMM
在线网址:
http://www.cbs.dtu.dk/services/TMHMM-2.0/
打开网站输入或提交fasta格式蛋白序列,选择有图模式,提交即可(如果提交大量序列建议选择One line per protein模式)。有图模式结果示例如下:
图中可以看出该蛋白有408个氨基酸,不存在跨膜区域(outside:1-408)。
TargetP
对于真核生物可以用TargetP预测蛋白质的亚细胞定位,TargetP在线网址为:
http://www.cbs.dtu.dk/services/TargetP/。
与SignalP相似的是提交序列为fasta格式蛋白,选择植物或非植物,点击提交。
TargetP示例预测结果如下,从结果看该蛋白包含信号肽(Loc为S),不定位在线粒体上,RC(可靠性等级)为1,为最可靠的预测。
若Loc为M,RC为1,表明该蛋白很可能定位在线粒体上。
PredGPI
用PredGPI可以预测GPI锚定蛋白,该软件只有在线版,网址为:
http://gpcr.biocomp.unibo.it/predgpi/pred.htm。
在预测界面输入或提交fasta格式蛋白序列,选择一般模式,点击Display即可完成预测。
从结果可以看出PredGPI提供四种预测结果,示例蛋白为Not GPI-anchored,表明该蛋白不是GPI锚定蛋白。
最后综合SignalP、TMHMM、TargetP和PredGPI的分析结果(存在信号肽、无跨膜区域、非定位在线粒体、非GPI锚定蛋白),即可预测出经典分泌蛋白或者植物病原菌的效应子(预测效应子可以考虑将蛋白长度限定在300 aa)。另外这些软件一般都对一次提交的蛋白序列数目或氨基酸数目有所限制,如果需要对大量序列进行预测,可以将其拆分成小文件。提交大量序列预测需要的时间会较长,可以在输入界面或运行界面输入个人邮箱,在运行完成后网站会以邮件的形式通知,结果展示仍然是网页形式,由于网页结果保存时间有限,因此在预测运行完成后需要及时保存结果。当然除了上述几款软件还有其他软件可以预测信号肽和跨膜区域,如phobius(http://phobius.sbc.su.se/),我们可以根据实际需求选择相应的工具进行分析。
参考文献
1. Jeffery L. Dangl & Jonathan D. G. Jones.,Plant pathogens and integrated defence responses to infection,Nature411, (2001).
2. Renier A.L. van der Hoorn and Sophien Kamoun.,From guard to decoy: a new model for perception of plant pathogen effectors, The Plant Cell20, (2008).
3. Kamoun S. A catalogue of the effector secretome of plant pathogenicoomycetes. Annu Rev Phytopathol. 44, (2006).
4. Diane G. O. Saunders, Joe Win, et al. Using hierarchical clusteringof secreted protein families to classify and rank candidate effectorsof rust fungi. PLoS One7, (2012).
5.Parham Haddadi,Lisong,Ma, et al.Genome-wide tranomic analyses provide insights into thelifestyle transition and effector repertoire of Leptosphaeriamaculans during the colonization of Brassica napus seedlings. Molecular Plant Pathology17, 1196-1210 (2016).
6. Zhang Liqing, Huang Xin, et al.Novel Fungal Pathogenicity and Leaf Defense Strategies Are Revealed by Simultaneous Tranome Analysis of Colletotrichum fructicola and Strawberry Infected by This Fungus. Front. Plant Sci. 9, (2018).
7. Petersen T N, Brunak S, Von H G, et al. SIGNALP4.0:discriminating signal peptides from transmembrane regions. Nature Methods, 2011, 8(10):785-6.
8. A. Krogh, B. Larsson, G. von Heijne, and E. L. L. Sonnhammer. Predicting transmembrane protein topology with a hidden Markov model: Application to complete genomes.Journal of Molecular Biology,305(3):567-580,2001.
9. Emanuelsson O, Nielsen H, Brunak S, von Heijne G. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence.J Mol Biol.2000 Jul 21;300(4):1005-16.
10. Pierleoni A, Martelli PL, Casadio R. PredGPI: a GPI anchor predictor. BMC Bioinformatics 9 (2008).